研究テーマ
生命分子システムの動的秩序形成と高次機能発現の仕組みの探究
キーワード
生命分子、動的秩序、高次機能、NMR
生命現象の特質は、システムを構成する多数の分子素子がダイナミックな離合集散を通じて秩序構造を形成し、外的環境との相互作用を行いつつ、自律的に時間発展していくことにあります。前世紀末期に勃興したオミクスアプローチは生命体を構成する分子素子に関する情報の網羅的集積を実現しました。しかしながら、それらの生命素子が自律的に柔軟かつロバストな高次秩序を形成するメカニズムを理解することは、これからの生命科学の重要な課題です。私たちは、生物学・化学・物理学の分野横断的な研究を通じて、内的複雑性を秘めた生命分子素子が動的な秩序を形成して高次機能を発現する仕組みを分子科学の観点から解き明かすことを目指しています。
さらに、生命分子システムのデザインルールを取り入れた人工自己組織化システムの創生に資することを目的とした研究も行っています。生命超分子集合体は、外部環境の変動や超分子集合体間のコミュニケーションを通じて時空間的発展を遂げています。生命分子システムの有するこうした特徴の本質を深く理解し、それを積極的に人工超分子系の設計に取り入れることは、分子科学におけるパラダイムシフトをもたらすものと考えています。
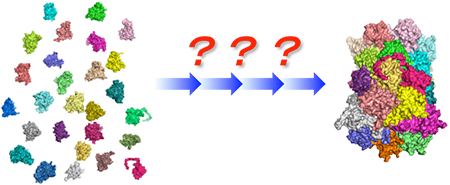
Formation of supramolecular machinery through dynamic assembly and disassembly of biomolecules.
参考文献
- K.Kato, S.Yanaka, and T.Yamaguchi, “The synergy of experimental and computational approaches for visualizing glycoprotein dynamics: Exploring order within the apparent disorder of glycan conformational ensembles”, Curr. Opin. Struct. Biol. 92, 103049 (2025)
- K.Kato, S.Yanaka, and H.Yagi, “Technical basis for nuclear magnetic resonance approach for glycoproteins”, Experimental Approaches of NMR Spectroscopy II (The Nuclear Magnetic Resonance Society of Japan ed.), Springer Nature Singapore, pp.169-195 (2025)
- H.Yagi, K.Takagi, and K.Kato, “Exploring domain architectures of human glycosyltransferases: Highlighting the functional diversity of non-catalytic add-on domains,” Biochim. Biophys. Acta –General Subjects, 1868, 130687 (2024)
- D. Koga, S.Kusumi, H.Yagi, and K.Kato, “Three-dimensional analysis of the intracellular architecture by scanning electron microscopy”, Microscopy, 73, 215-225 (2024)
- K.Kato and H.Yagi, “Current status and challenges in structural glycobiology,” Trends in Carbohydrate Research, 15, 38-46 (2023).
- Yagi-Utsumi, M. and Kato, K., “Conformational variability of amyloid-β and the morphological diversity of its aggregates,” Molecules 27, 4787 (2022).

