サイト内検索
分子研について
研究情報
共同利用案内
- 共同研究・施設利用案内
- 申請概要
- 共同研究受入研究室・利用装置一覧
- WEB申請
大学院
- 大学院教育
- 大学院案内
- 受験生向け情報
- 他大学の学生の受け入れ
- 在校生向け情報
研究・研究者
研究グループ詳細
グループリーダー

生体分子マシンの機能発現ダイナミクスの解明とその制御
理論生物物理、分子モーター、トランスポーター、バイオエナジェティクス、分子シミュレーション
細胞で働く分子モーターやトランスポーターなど生体分子マシンは、機能する際に構造を変化させることが知られています。例えば、ミトコンドリアでATPを合成するATP合成酵素は、化学エネルギーを用いて回転する分子モーターです。細胞膜を超えて基質分子を輸送するトランスポーターは、膜に対して内側に開いた構造と外側に開いた構造との間で構造変化することで、基質輸送を行っています。このように自然が作り上げた精巧かつダイナミックなナノマシンが働く仕組みを原子・分子レベルで解明し、その知見に基づいた機能制御を目指しています。
私たちは、生体分子マシンが機能する瞬間の動き「機能発現ダイナミクス」を、原子・分子レベルでコンピュータ上に再現して「見る」ことで、その仕組みを理解したいと思っています。しかしながら、これは容易なことではありません。なぜなら、生体分子マシンは、巨大な分子である上に、機能する時間スケールはミリ秒以上と(分子スケールにしては)遅いからです。数十万原子以上からなる巨大な系のミリ秒時間スケールの動きをシミュレーションするのは通常の手法では困難です。そこで、動く瞬間を切り出してシミュレーションする手法や、複数原子をまとめて粗視化する手法などを用いて、機能する瞬間の動きを捉えようとしています。
私たちはこれまで、細胞内の主なエネルギー供給源であるATP合成酵素や1) 、細胞内のイオン濃度を調節するトランスポーターNa+/H+交換輸送体2,3)に取り組んできました。Na+/H+交換輸送体においては、イオン輸送が起こる瞬間の動きをシミュレーションで捉えて、イオン輸送を制御している重要な相互作用を同定しました。この相互作用を実験的に調節することで、イオン輸送速度を2倍以上速くすることに成功しました。このように、シミュレーションで解明した生体分子マシンの仕組みに基づいた機能制御に挑戦していきます。
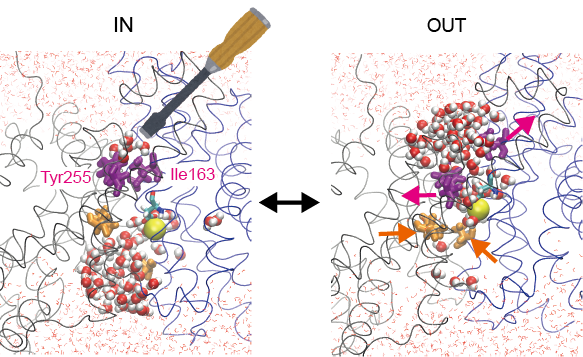
Control of the transporter function based on mechanism elucidated by simulation.