サイト内検索
分子研について
研究情報
共同利用案内
- 共同研究・施設利用案内
- 申請概要
- 共同研究受入研究室・利用装置一覧
- WEB申請
大学院
- 大学院教育
- 大学院案内
- 受験生向け情報
- 他大学の学生の受け入れ
- 在校生向け情報

古代ギリシアの哲学者アリストテレスの著書「自然学」には時間に関する次のような記述がある。
さて、それゆえに、われわれが「今」を、運動における前のと後のとして
でもなく、あるいは同じ今だが前の今の或る〔終りの〕部分と後の今の或
る〔初めの〕部分としてでもなしに、一つのものとして知覚する場合には、
そこにはなんらの〔意識上の〕運動もないわけだから、なんらの時間も経
過したとは思われない。これに反して、前と後を知覚する場合には、われ
われはそこに時間があると言う。というのは、時間とはまさにこれ、すな
わち、前と後に関しての運動の数であるから 。
アリストテレス全集3[1]より引用
運動の数をもって時間となすという捉え方である。鹿威し、日時計、クォーツ時計、そして原子時計̶̶精度も種類も様々であるが、確かに古代より我々人類はこの考え方のもとで時間という概念を作り出し活用してきた経緯がある。本稿では、時を刻むタンパク質(直径約10 nm)が存在していること、そしてそのタンパク質の構造や運動に地球の自転周期にあたる24 時間の時定数が書き込まれていることを紹介し、最後にこの分野の展望について述べたい。
生物時計は次の3 つの性質を備えている。
①恒常的条件において約24 時間(概日)周期で発振する(自律的発振)。
②周期(周波数)が温度に依存しない(温度補償性)。
③環境変動に応じて振動の位相を変化させて同調することができる(同
調能)。
図1 は生物時計の基本骨格の概念図である。コアとなる振動体はそれ単独であっても概日周期で発振し(性質①)、その周期は温度に対して補償されている(性質②)。光や温度といった外界の周期的な変動が入力系を介して振動体へと伝わり、これによって振動体の位相が前後にシフトする(性質③)。振動体の位相は時刻情報として出力系を介して伝搬され、リズミックな遺伝子発現を誘導する。これにより代謝や行動レベルで概日リズムが生じる。
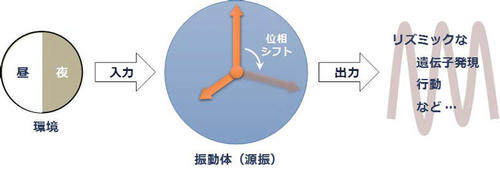 図1 生物時計の基本骨格の概念図。図は文献(14)より改変。
図1 生物時計の基本骨格の概念図。図は文献(14)より改変。
リズミックな現象を扱う際には何が本質であるかに常に注意を払う必要がある。周期的な反応であるため、ときに原因と結果を区別することが容易でないためである。また、リズムが観察されたときに、それが出力を介したリズムであるのか(クォーツ時計の針)、それとも振動体(水晶の発振周期)を捉えているのか、単純なリズム観察から判定することはできない。振動の源(源振)を捉えているのであれば(図1)、入力系と出力系を取り除いた(隔絶された)状況でも振動は継続するはずである。
シアノバクテリアの生物時計に関する研究は、その源振の所在を突き詰める方向に進展してきた。1998 年に時計遺伝子(kai 遺伝子)が同定され[2]、その当事は、kai 遺伝子発現のネガティブ・フィードバック制御の結果としてリズムが生成されるものと解釈された。これは転写時計と呼ばれるモデルであり、今日もシアノバクテリア以外の高等生物では主要な学説となっている。2005年、近藤らはkai 遺伝子発現やその制御が源振ではなく、kai 遺伝子をもとに作られた3 種類のKai タンパク質(KaiA、KaiB、KaiC)をアデノシン三リン酸(ATP)と試験管内で混合するだけで、安定な概日リズムが生じることを発見した(Kai タンパク質時計)[3]。我々は、その後の研究を通じて[4-8]、KaiC が時計のコアであることを突き止めた。
KaiC は結合したATP をアデノシン二リン酸(ADP)とリン酸(Pi)に加水分解する機能(ATPase)を有する。図2 に示すように、KaiA とKaiB が共存する環境では、加水分解の反応速度(ATPase活性)が24 時間周期で自律的に発振する(ATP が存在する限り振動は何週間~月でも継続する)。KaiA とKaiB が存在しないとKaiC のATPase 活性が安定に発振することは(今のところ)ないが、まったく発振しないというわけでもないことに我々は気が付いた[7]。図2 が示すように、KaiC 単独であってもATPase 活性は減衰振動を示し、制御工学に基づいた解析(2 次の伝達関数を仮定)を行ったところ減衰振動成分が0.91 d-1 の固有振動数(ω)を有することがわかった。これは地球の自転とほぼ同じ頻度(1 回/日)で運動する“何か” がタンパク質分子であるKaiC に内包されていることを示している。KaiA とKaiBとKaiCが共存すると自律的かつ安定的に発振、しかしKaiAとKaiB のみでは減衰型の振動さえ示さない̶̶これらのことを考慮すると、KaiC こそが源振であり、そこにエンコードされているωがシアノバクテリアの生物時計の針を進める速度を決定する因子であるといえよう。
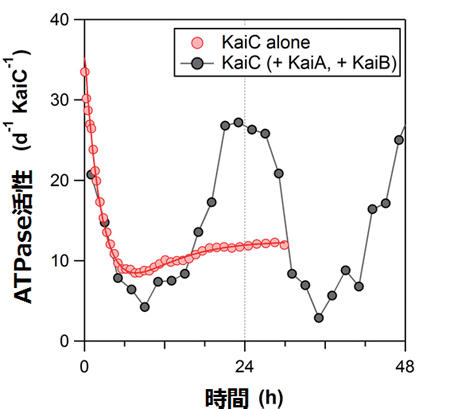
図 2 リズミックなKaiC のATPase 活性。 図は文献(7)より改変。
興味深いことに、ω の値にはKaiCのATPase 活性(N 末端ドメイン)と高い相関が見られた[7]ーーアミノ酸変異によってKaiC 単独でのATPase 活性が2 倍に向上すると、ω(図3C)や転写時計の振動数(図3B)も2 倍になる。KaiC のATPase 活性(12 d-1)は、よく知られているモータータンパク質(103~ 107 d-1)に比べて極端に低く、かつ温度補償制御されている(Q10 = 1.0 ~1.1、生物時計の第二の性質)。これらは、ATPase 活性を化学的に制御しているタンパク質構造や動態が物事の本質であることを示している。
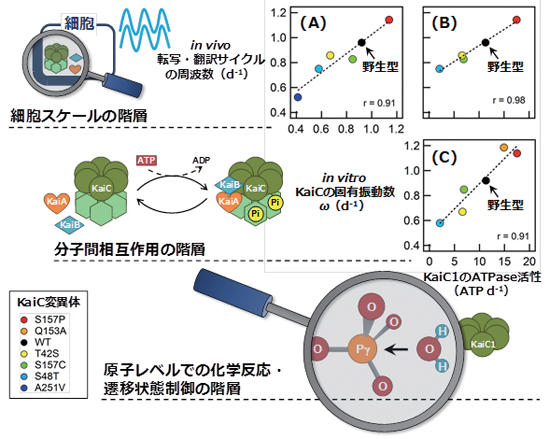 図 3 シアノバクテリア概日時計システムの貫階層性。(A) 転写時計の周波数とKaiC のωの相関プロット。(B) 転写時計の周波数とKaiC のATPase 活性の相関プロット。(C)KaiC のω
図 3 シアノバクテリア概日時計システムの貫階層性。(A) 転写時計の周波数とKaiC のωの相関プロット。(B) 転写時計の周波数とKaiC のATPase 活性の相関プロット。(C)KaiC のω
とATPase 活性の相関プロット。※遺伝子組換え技術を用いて、KaiC の狙った場所にアミノ酸変異を導入し、その変異型タンパク質を細胞内で発現させることができる。変異の導入箇所によってシアノバクテリアの生物時計システムの周期が短くなったり(短周期型変異)、長くなったりする(長周期型変異)。KaiC 変異体を精製して試験管内でωを実測してみると、細胞(転写時計)のリズムの振動数と良い一致が見られた(パネルA)。図は文献(7) より改変。
構造解析に着手してから9 年後の2015 年、KaiC のATP 加水分解反応に関する構造基盤を発表した(7)。加水分解とはその名の通り、ATP の末端(γ)リン酸基のリン原子(Pγ)に水分子(W1)を反応させ、ADP とPi に分解する過程である。よって反応効率を考えるうえで、ATP に対するW1 の空間配置が重要となる。ここでは図4 の模式図をもとに、Pγ とW1 の酸素原子(OW1)の距離(dPγ-OW1)、Pγ から隣接酸素原子(O3β) とOW1 を見込む角度(∠ O3β-Pγ-OW1)、この2 つのパラメータに焦点を絞って説明する。ATP 加水分解がSN2 型の求核置換反応であるとすると、OW1 がPγ-O3β の結合軸に沿うようにPγ に接近(求核的攻撃)するのが最も至適な反応経路となる(∠ O3β-Pγ-OW1 = 180˚)。しかしながら、KaiC のOW1 はdPγ-OW1 = 3.8~4.6Å、∠O3β-Pγ-OW1 = 141~154˚ の位置に確認され、他のATPase よりもATPから遠くかつ反応に不利な配置にあることがわかる。実験的に決定したタンパク質構造をもとに理論化学的な計算を行ったところ、立体障害のためにW1 は加水分解に適した位置(図4 の十字、dPγ-OW1 = 3.0Å、∠O3β-Pγ-OW1 =180˚)には全く侵入できず、逆に、それほど安定化されていないため容易に他の場所へ散逸してしまうことがわかった。これが、KaiC のATPase 活性を極端(24 時間周期にマッチする程度)に低くしている原因の一つである。活性の高いATPase であっても、W1 が求核攻撃する際の活性化エネルギーは11~ 17 kcal mol-1 に達することが知られている[9-11]。KaiC のATPase がより大きな活性化エネルギーを要することは想像に難くない。
著しい低活性(12 d-1)とはいえ、W1 の進入を防ぐ立体障害は1 日に12回程度解消され、加水分解へのゲートが開かれるはずである。我々はADP 結合状態の結晶構造解析に成功し、加水分解前後での構造比較を通して立体障害の解消過程の一端を解明した。立体障害を取り除くためには、3 本のαヘリックスを大規模に移動させつつ、同時にSer145-Asp146 間のペプチド結合をcis 型からtrans 型へ異性化させる必要がある(図5)。ポリペプチド鎖の異性化はタンパク質分子の運動の中でもかなり遅い部類にあたる[12]。局所的な異性化であったとしても14 ~ 16kcal mol-1 の活性化エネルギーが必要になることが判明した。これが、KaiC の
ATPase を低活性化させている別の要因である。
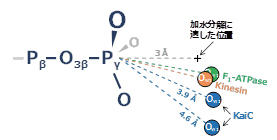
図4 ATP と加水分解水(W1)の位置関係を示した模式図。図は文献(7) より改変。
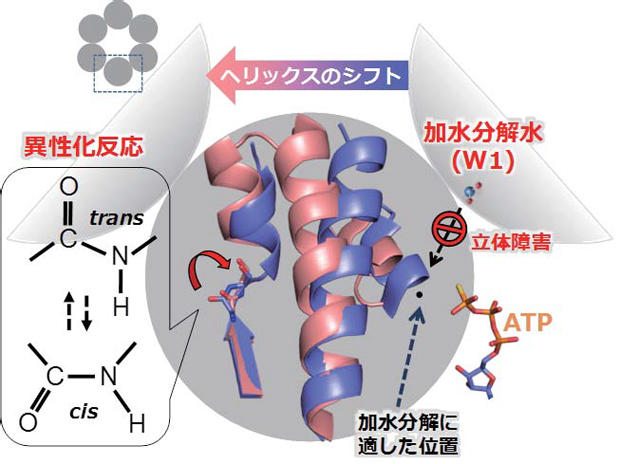
図5 KaiC のATP加水分解反応を遅くしている構造的要因。図は文献(7) より改変。
「W1 をPγ に接近(>11 ~ 17 kcal mol-1)」させるためには、「αヘリックス群をシフト(? kcal mol-1)」させつつ、同時に「ペプチド結合を異性化(>14~ 16 kcal mol-1)」させなくてはならない。これらのイベントが反応座標上でどれくらい近接(共役)しているか
については未解明な点が残されているため、今のところ正味の活性化エネルギーを正しく計算することは難しい。303.15 K 下で1012 s-1 の頻度因子を想定すると、12 d-1(0.5 h-1)の反応速度を説明する活性化エネルギーは約22kcal mol-1 と見積もられる。これは一連の構造転移が個別というよりも一体となって進行することにより、非凡なまでに低く安定したATPase 活性が実現されていることを示唆する。
我々は2012年に分子研で研究グループを立ち上げ、「①概日周期での発振」を取っ掛かりに24 時間周期を定める遅さの根源へと研究を展開し、今後は「②温度補償性」そして「③同調能」に関する理解を深め、最終的には時間を扱う生物学(時間生物学)を分子科学の観点から極めたいと考えている。①については相応の手ごたえが感じられつつあるものの、生物時計の理解が3 大性質(①~③)を漏れなく同時に説明することだとすると、我々の到達点はまだまだ道半ばということになる。
次の目標は①と②を同時に説明することである。遅い化学反応は高い活性化エネルギーを有することが多く、温度依存的に加速されるのが一般的である。時計の針が一定の速度で進むよう、周期を定めているKaiC のATPase は厳密に温度補償制御されている。究極的に遅い化学反応を厳密に温度補償制御するために消費されるエネルギーがほんの僅か(一日あたり12 ATP)であることにも首をかしげずにはいられない。いつの日かこの自然の叡智を理解し、そう遠くない未来にモノづくりや環境低負荷型の情報処理に活かせないものであろうか。
向山厚博士、古池美彦博士、阿部淳博士をはじめとする研究グループの同僚、そして近藤孝男博士(名古屋大学)、山下栄樹博士(大阪大学)、斎藤真司博士(分子科学研究所 理論・計算分子科学領域)、森俊文博士(分子科学研究所理論・計算分子科学領域)をはじめとする多くの共同研究者に恵まれ、今日まで研究活動を継続することができました。この場をお借りして御礼申し上げます。誌面の都合上、また読み易さを優先するために科学的な説明を一部省略して記述しました。詳細については別に記した総説(13-15) などを参照して頂ければ幸いです。
[1] アリストテレス全集3 自然学、出隆/岩崎允胤訳、岩波書店、1968年.
[2] M. Ishiura, S. Kutsuna, S. Aoki, H. Iwasaki, C.R. Andersson, A. Tanabe, S.S. Golden, C.H. Johnson, and T. Kondo, Expression of a gene cluster kaiABC as a circadian feedback process in cyanobacteria, Science, 281, 1519-1523 (1998).
[3] M. Nakajima, K. Imai, H. Ito, T. Nishiwaki, Y. Murayama, H. Iwasaki, T. Oyama, and T. Kondo, Reconstitution of circadian oscillation of cyanobacterial KaiC phosphorylation in vitro, Science, 308, 414-415 (2005).
[4] S. Akiyama, A. Nohara, K. Ito, Y. Maéda, Assembly and Disassembly Dynamics of the Cyanobacterial Periodosome, Molecular Cell, 29, 703–716 (2008).
[5] Y. Murayama, A. Mukaiyama, K. Imai K, Y. Onoue, A. Tsunoda, A. Nohara, T. Ishida, Y. Maéda, K. Terauchi, T. Kondo, and S. Akiyama S, Tracking and visualizing the circadian ticking of the cyanobacterial clock protein KaiC in solution, The EMBO Journal, 30, 68-78 (2011).
[6] A. Mukaiyama, M. Osako, T. Hikima, T. Kondo, and S. Akiyama S, A protocol for preparing nucleotide-free KaiC monomer, BIOPHYSICS, 11, 79-84 (2015).
[7] J. Abe, T. B. Hiyama, A. Mukaiyama, S. Son, T. Mori, S. Saito, M. Osako, J. Wolanin, E. Yamashita, T. Kondo, and S. Akiyama, Atomic-scale Origins of Slowness in the Cyanobacterial Circadian Clock, Science, 349, 312-316 (2015).
[8] Y. Furuike, J. Abe, A. Mukaiyama, and S. Akiyama, Accelerating in vitro studies on circadian clock systems using an automated sampling device, Biophysics and Physicobiology, 13, 235-241 (2016).
[9] M.J. McGrath, I.F. Kuo, S. Hayashi, and S. Takada, Adenosine triphosphate hydrolysis mechanism in kinesin studied by combined quantum-mechanical/molecular-mechanical metadynamics simulations, J. Am. Chem. Soc., 135, 8908-8919 (2013).
[10] B.L. Grigorenko, A.V. Rogov, I.A. Topol, S.K. Burt, H.M. Martinez, and A.V. Nemukhin, Mechanism of the myosin catalyzed hydrolysis of ATP as rationalized by molecular modeling, Proc. Natl. Acad. Sci. USA, 104, 7057-7061 (2007).
[11] S. Hayashi, H. Ueno, A.R. Shaikh, M. Umemura, M. Kamiya, Y. Ito, M. Ikeguchi, Y. Komoriya, R. Iino, and H. Noji, Molecular mechanism of ATP hydrolysis in F1-ATPase revealed by molecular simulations and single-molecule observations, J. Am. Chem. Soc., 134, 8447-8454 (2012).
[12] M. Karplus, Aspects of protein reaction dynamics: deviations from simple behavior, J. Phys. Chem. B, 104, 11-27 (2000).
[13] 向山厚,阿部淳,孫世永,秋山修志, タンパク質の化学反応が細胞内の時を計る, 実験医学, 33, 3119-3122 (2015).
[14] 秋山修志, 時間生物学と放射光科学の接点, 放射光, 29, 56-63 (2016).
[15] 秋山修志, 概日時計因子の構造や動態を調べる意義とは? , 生物物理, 56, 266-270 (2016).